我院姚成果课题组和厦门大学叶从庭课题组合作发表U1 snRNP telescripting最新分子机制模型
U1 snRNP复合体(U1 snRNA, U1A, U1C, U1-70K以及7个Sm蛋白)是细胞内迄今为止研究得最多,且含量最为丰富的蛋白复合体(约100万个拷贝/细胞)之一。其经典的分子生物学功能是生化教科书中描述的真核生物pre-mRNA splicing功能。2010年,Gideon Dreyfuss实验室发现U1 snRNP具有维持新生全长mRNA转录的新功能,并命名为U1 snRNP telescripting。鉴于U1新功能在真核细胞内存在的普遍性以及影响基因的广泛性, 其普适性分子机制的解析是当前mRNA共转录加工分子机制研究中的重难点问题。

5月22日,我院干细胞中心姚成果课题组与厦门大学叶从庭课题组合作在JBC期刊(Journal of Biological Chemistry, 生化领域权威期刊)发表了题为”The U1 antisense morpholino oligonucleotide (AMO) disrupts U1 snRNP structure to promote intronic PCPA modification of pre-mRNAs”的论文,报道了针对领域内这一重难点问题的最新研究成果。这是该课题组近年在mRNA长度调控领域的又一进展(2017,NAR; 2018,2019,2021,2022,RNA Biol.; 2020,BBRC)。
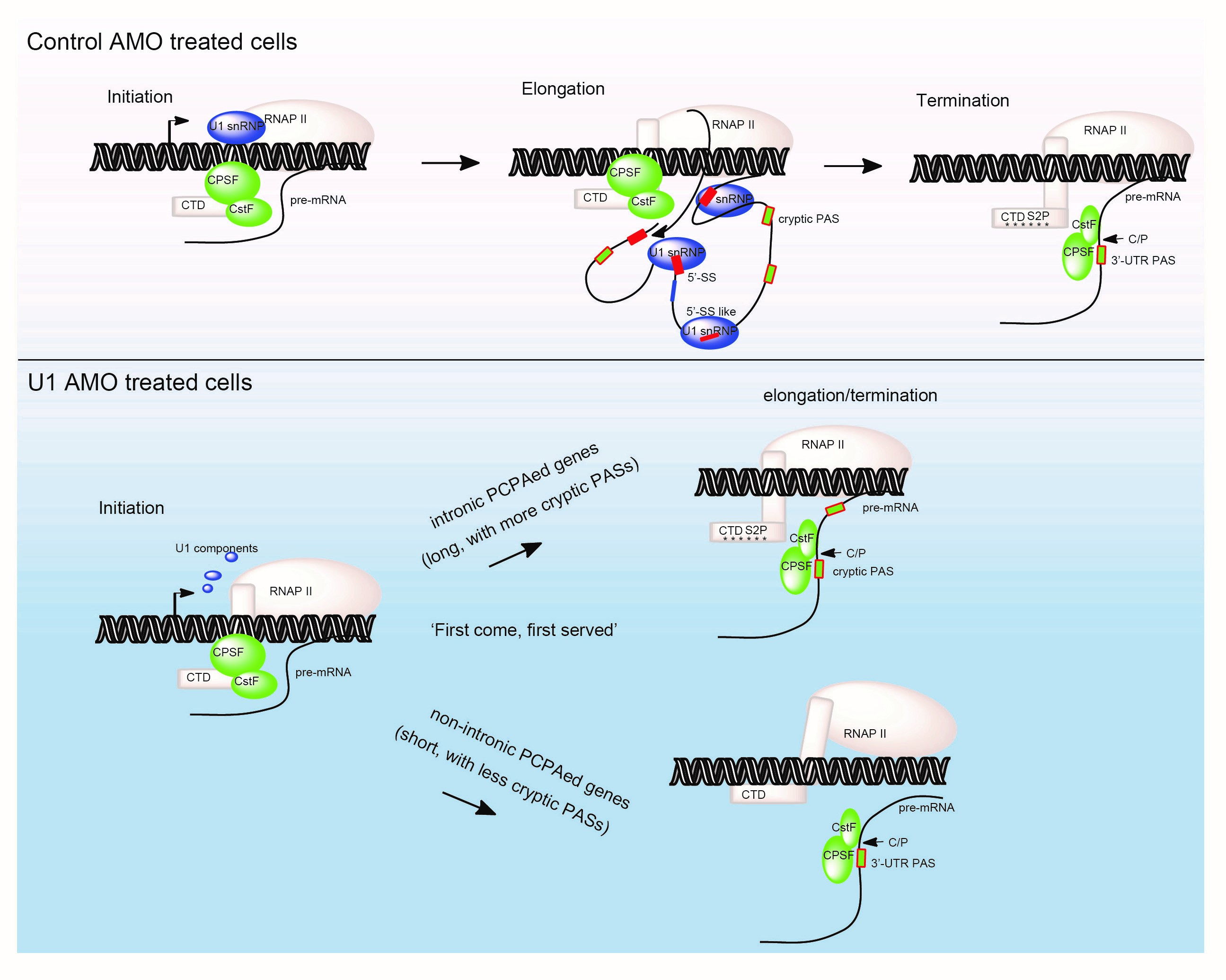
他们首先在富含U1 snRNP的HeLa细胞核抽提物中加入对照和U1 AMO,通过Co-IP, western blot发现后者可以破坏U1 snRNP复合体中的蛋白-蛋白相互作用,并进一步通过gel shift assay发现U1 AMO可以直接破坏U1-70K与U1 snRNA的相互作用。这一新的发现为U1 AMO体外破坏U1 snRNP结构提供了直接的证据。进一步细胞内实验发现,当细胞被转染U1 AMO后,相比对照,U1 snRNP的结构不仅遭到破坏,同时还造成了部分U1-70K和U1C的明显转运出核。这种整体性的U1 snRNP的结构破坏影响了U1 snRNP与RNAPII的结合,以及RNAPII/RNAPII CTD-Ser2/Ser5在活跃转录基因组上的分布。
进一步针对核心mRNA3'末端加工因子CPSF/CstF的ChIP-seq和iCLIP-seq实验表明,U1 AMO处理条件下,CPSF/CstF在共转录加工过程中被提前招募到染色质基因组上,并与内含子内polyA site相互作用,而造成pre-mRNA变短。
总之,该研究挑战并更新了2019年Dreyfuss实验室在Mol Cell杂志所发表的U1-CPAF模型,主要创新点有:(1)以更为扎实的直接的数据证明,U1 AMO在体外和体内都直接破坏了U1 snRNP的结构,挑战了U1-CPAF模型中认为U1 AMO处理不影响U1 snRNP的结构的观点;(2)在共mRNA共转录加工过程中,核心3’末端加工因子CPSF/CstF与内含子polyA site的相互作用是理解U1 snRNP telescripting机制的另一关键点。
我院研究生冯秋敏和厦门大学的研究生林泽瑾为论文共同第一作者,厦门大学的叶从庭副教授是论文的共同通讯,我院姚成果副教授是最后主要通讯作者。该研究得到了多项基金,包括国家自然科学基金面上项目和广东省自然科学基金面上项目的资助。
原文链接:https://www.sciencedirect.com/science/article/pii/S0021925823018823



