Activities
New publishment in Nature Structural & Molecular Biology 在NSMB新发表文章一篇

https://doi.org/10.1038/s41594-023-01066-9
染色质结构的动态折叠在真核生物中具有重要的生物学功能【1-3】。例如染色质在大尺度层级被划分为转录活跃的A区室(A compartment)和转录抑制的B区室(B compartment)【4,5】。然而,在染色质三维结构【6,7】和转录【8,9】的关系研究中,大多是基于传统方法在细胞群体中获取的平均信息,导致每一个细胞的异质性被掩盖,使得我们不能很好地探究染色质三维结构和转录之间的关系【10,11】。为了克服单细胞信息被掩盖的问题,目前已经出现了许多单细胞三维基因组检测技术【12,13】和单细胞RNA检测技术【14,15】。尽管这些方案实现了单细胞信息的展示,但由于染色质三维结构和转录的数据产自不同的单细胞,仍旧无法准确地反映两者之间的关系。因此,我们亟需一种新的检测技术。
2023年8月14日,中山大学丁俊军课题组在Nature Structural & Molecular Biology发表了题为Simultaneous profiling of chromatin architecture and transcription in single cells的研究成果。该研究建立了一套基于二代测序方法开发的单细胞多组学检测技术scCARE-seq(Single Cell Chromatin Architecture and mRNA Expression),实现了在单个细胞中同时检测染色质三维结构和转录表达。
scCARE-seq技术流程概括来说,首先在完整的细胞核中进行mRNA反转录、基因组消化和连接,然后通过流式将单个细胞分选到96孔板独立的孔中进行裂解,并在每个孔中加入Tn5转座酶进行片段化。接下来,对每个单细胞文库进行预扩增并一分为二。最后,使用特异性的引物对DNA和RNA文库分别扩增后测序。
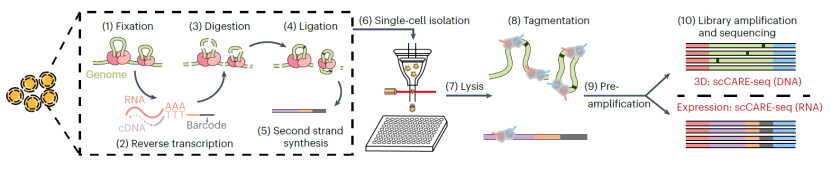
图1. scCARE-seq流程示意图
尽管在细胞周期中,染色质三维结构和转录的变化已经在群体和单细胞水平上被分别捕获,但它们的调控关系却知之甚少。首先,研究人员根据scCARE-seq中的染色质三维结构信息,将每个细胞分配到细胞周期的不同阶段。随后,通过进一步引入相应单细胞的基因表达数据,研究人员在小鼠胚胎干细胞中全局水平发现了E-P相互作用与转录之间正相关的周期性变化(图2)。同时,研究人员还观察到了部分特定细胞周期差异基因的上下调与E-P相互作用的增减相关。这表明scCARE-seq具有促进细胞周期中染色质三维结构与转录之间动态调控关系的研究的能力。
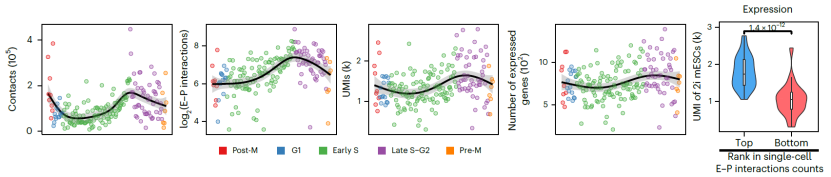
图2.小鼠胚胎干细胞在细胞周期中的染色质三维结构和转录水平的动态变化
中山大学中山医学院的博士生曲佳乐和博士后孙隽为本文的共同第一作者。中山大学丁俊军教授、暨南大学范丽丽老师和四川大学华西生物医学大数据中心曾筱茜副教授为该论文的共同通讯作者。
参考文献:
1. Bonev, B. et al. Multiscale 3D Genome Rewiring during Mouse Neural Development. Cell 171, 557-572.e524 (2017).
2. Du, Z. et al. Allelic reprogramming of 3D chromatin architecture during early mammalian development. Nature 547, 232-235 (2017).
3. Lupiáñez, Darío G. et al. Disruptions of Topological Chromatin Domains Cause Pathogenic Rewiring of Gene-Enhancer Interactions. Cell 161, 1012-1025 (2015).
4. Lieberman-Aiden, E. et al. Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science 326, 289-293 (2009).
5. Dixon, J.R. et al. Chromatin architecture reorganization during stem cell differentiation. Nature 518, 331-336 (2015).
6. Rao, Suhas S.P. et al. A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell 159, 1665-1680 (2014).
7. Kempfer, R. & Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics 21, 207-226 (2020).
8. Mortazavi, A., Williams, B.A., McCue, K., Schaeffer, L. & Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods 5, 621-628 (2008).
9. Marioni, J.C., Mason, C.E., Mane, S.M., Stephens, M. & Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res 18, 1509-1517 (2008).
10. Schwartzman, O. & Tanay, A. Single-cell epigenomics: techniques and emerging applications. Nature Reviews Genetics 16, 716-726 (2015).
11. Wen, L. & Tang, F. Single-cell sequencing in stem cell biology. Genome Biology 17, 71 (2016).
12. Tan, L., Xing, D., Chang, C.-H., Li, H. & Xie, X.S. Three-dimensional genome structures of single diploid human cells. Science 361, 924-928 (2018).
13. Ulianov, S.V., Tachibana-Konwalski, K. & Razin, S.V. Single-cell Hi-C bridges microscopy and genome-wide sequencing approaches to study 3D chromatin organization. Bioessays 39 (2017).
14. Tang, F. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods 6, 377-382 (2009).
15. Svensson, V., Vento-Tormo, R. & Teichmann, S.A. Exponential scaling of single-cell RNA-seq in the past decade. Nature Protocols 13, 599-604 (2018).
